
近日,我院王亚楠教授与合作者在中科院一区Top期刊《Microbiology Spectrum》发表了题为“Genomic analysis of almost 8,000 Salmonella genomes reveals drivers and landscape of antimicrobial resistance in China”的研究论文。该研究整合实验室已测序的1962个沙门菌基因组和6187个public数据,构建了迄今为止中国规模最大、综合性、高质量、开源的中国Local沙门菌基因组数据库(CLSGDB v2)。基于含有164个沙门菌血清型和295个序列型的CLSGDB v2,研究人员绘制并解析了沙门菌耐药基因、毒力基因和可移动遗传元件组图谱和时空动态特征。此外,还发现气候、社会和经济因素与沙门菌耐药性显著正相关。
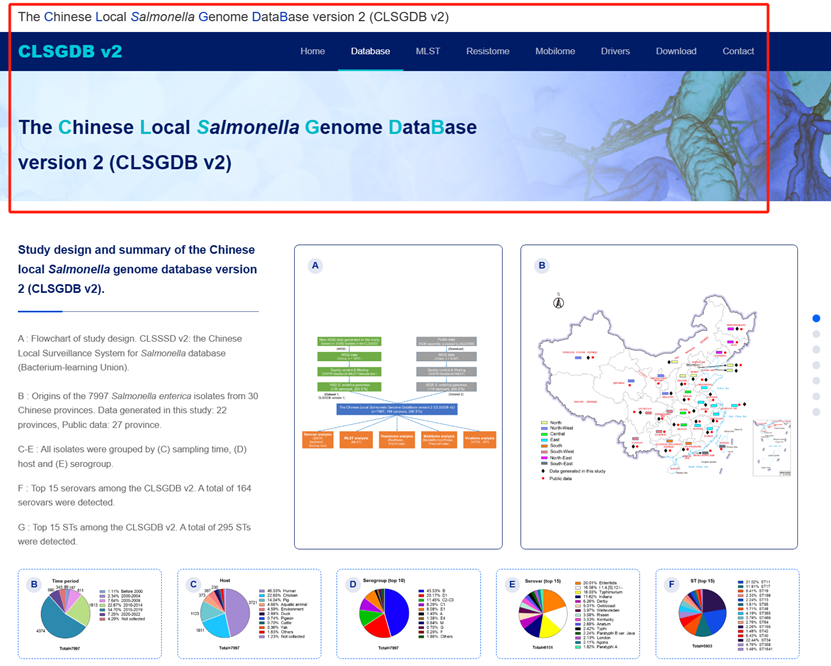
研究整合得到的中国沙门菌基因组集合数据(CLSGDB v2)存储于中国科学院微生物研究所国家微生物科学数据中心(https://nmdc.cn/clsgdbv2),供公开访问、获取和下载使用。
王亚楠教授为论文的第一作者,上海市疾病预防控制中心副主任技师许学斌为论文的共同第一作者。中国科学院微生物研究所高福院士和河南农业大学、龙湖现代免疫实验室张改平院士为论文的共同通讯作者。研究得到了国家自然科学基金、国家重点研发计划、河南省重大科技专项、河南农业大学高层次人才启动基金、中国科学院战略性先导科技专项、北京市科技计划等项目的资助。研究得到了国家微生物科学数据中心在数据存储、可视化和维护方面的支持和帮助。
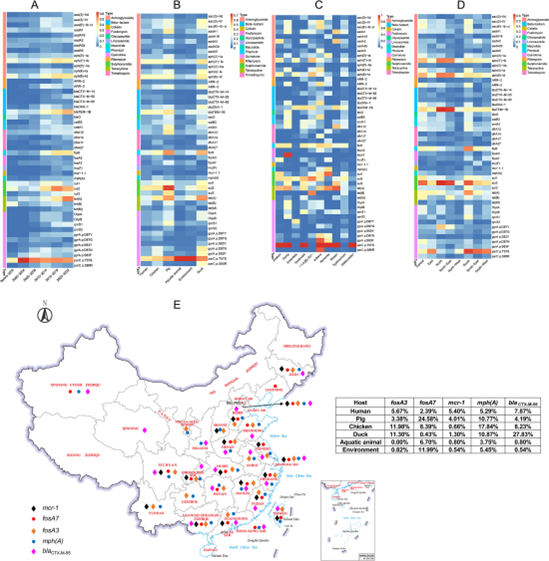
最后,研究团队将构建的综合性、高质量、目前中国规模最大的沙门菌基因组数据库坂本2(CLSGDB v2)作为一个开放访问的数据库发布(访问网址:https://nmdc.cn/clsgdbv2),从而可以帮助监测研究164个沙门菌血清型和295个序列型。
相关阅读:
中国Local沙门菌基因组数据库版本2(CLSGDB v2)的构建和应用是团队前期研究工作的延续。王亚楠教授前期与合作者基于>3.5万株沙门菌和近2000个基因组数据揭示了2006-2019年我国沙门菌优势血清型及抗菌素耐药性的动态变化,发现猪源ST34和鸡源ST19鼠伤寒沙门菌分别与引起儿童和成人感染的菌株有密切的相关性,在7个血清型中首次发现12个新的序列型,首次在我国人源粪便样本中分离出替加环素耐药的兰道夫沙门菌,在质粒上检测到tet(X4)基因,为公共卫生决策者优先考虑食源性疾病和食品安全干预措施提供了重要的参考依据。
参考文献:
1.Wang Y#, Xu X#, Zhu B, Lyu N, Liu Y, Ma S, Jia S, Wan B, Du Y, Zhang G*, Gao GF*. Genomic analysis of almost 8,000 Salmonella genomes reveals drivers and landscape of antimicrobial resistance in China. Microbiology Spectrum. 2023; DOI: https://doi.org/10.1128/spectrum.02080-23.
2.Wang Y#, Liu Y#, Lyu N, Li Z, Ma S, Cao S, Pan Y, Hu Y, Huang H, Gao GF*, Xu X* on behalf of the Bacterium-learning Union, & Zhu B*. The temporal and spatial dynamics of antimicrobial-resistant Salmonella enterica and predominant serovars in China. National Science Review. 2023; 10(3): nwac269.
3.Wang Y#, Liu F#, Xu X#, Huang H, Lyu N, Ma S, Chen C, Mao M, Hu Y, X Song, Li J, Pan P, Wang A, Zhang G, Zhu B, Gao GF*. Detection of plasmid-mediated tigecycline resistance gene tet(X4) in a Salmonella enterica serovar Llandoff isolate. Infectious Microbes & Diseases. 2021; 3, 198-204.
